Processamento de Dados
O processamento de dados é sem dúvida a etapa mais importante e que mais consome tempo em um fluxo de análises. Nesta seção vamos abordar algumas funções básicas do tidyverse que auxiliam bastante na realização de tarefas triviais de edição de dados.
1 Combinando Datasets
Em muitas situações é comum que seja necessário combinar múltiplas fontes de dados. Em melhoramento animal por exemplo é comum que informações dos mesmos animais estejam dispersas por arquivos de dados individuais.
Vamos dar uma olhada nos datasets que temos disponíveis? Se lembram como fazer?
Os arquivos NELPN.csv, NELP365.csv e NELP550.csv possuem dados de 50 animais com respectivas medidas biométricas para Peso ao Nascimento (PN), Peso a um ano (P365) e Peso ao Sobreano (P550). Estes dados foram simulados com base em animais da raça Nelore.
Uma vez que os mesmos animais estão nos três conjuntos de dados, nosso objetivo é unir estes arquivos em um único conjunto de dados. Vamos fazer isso de duas maneiras diferentes.
1.1 Modo Básico
Carregamos os arquivos individualmente cada um em um objeto
Usamos as funções de join do
tidyversepara unir os objetos
Rows: 50 Columns: 2
── Column specification ────────────────────────────────────────────────────────
Delimiter: ","
chr (1): ID
dbl (1): PN
ℹ Use `spec()` to retrieve the full column specification for this data.
ℹ Specify the column types or set `show_col_types = FALSE` to quiet this message.Rows: 50
Columns: 2
$ ID <chr> "RV143", "YB821", "QA008", "WT997", "IT185", "PD544", "SL754", "MC3…
$ PN <dbl> 36.04, 31.09, 32.33, 28.35, 25.06, 37.53, 33.23, 24.19, 30.05, 37.9…Rows: 50 Columns: 2
── Column specification ────────────────────────────────────────────────────────
Delimiter: ","
chr (1): ID
dbl (1): P365
ℹ Use `spec()` to retrieve the full column specification for this data.
ℹ Specify the column types or set `show_col_types = FALSE` to quiet this message.Rows: 50
Columns: 2
$ ID <chr> "RV143", "YB821", "QA008", "WT997", "IT185", "PD544", "SL754", "M…
$ P365 <dbl> 211.15, 194.81, 186.59, 192.42, 202.37, 189.50, 197.18, 160.95, 2…Rows: 50 Columns: 2
── Column specification ────────────────────────────────────────────────────────
Delimiter: ","
chr (1): ID
dbl (1): P550
ℹ Use `spec()` to retrieve the full column specification for this data.
ℹ Specify the column types or set `show_col_types = FALSE` to quiet this message.Rows: 50
Columns: 2
$ ID <chr> "RV143", "YB821", "QA008", "WT997", "IT185", "PD544", "SL754", "M…
$ P550 <dbl> 290.43, 304.21, 305.59, 285.34, 331.91, 273.09, 295.91, 335.95, 2…A função glimpse é bastante útil pra nos mostrar uma prévia dos nossos dados.
Agora que nossos arquivos já foram carregados, podemos fazer a combinação (merge) dos dados. Para este tipo de operação no qual temos os mesmos animais e variáveis medidas em arquivos separados, usaremos a ID de cada animal como uma chave de identificação entre os conjuntos de dados, desta maneira sabemos que teremos cada observação de cada animal corretamente alocada no nosso novo conjunto de dados.
No nosso caso usaremos a função inner_join do dplyr. Esta função recebe dois conjuntos de dados e combina as observações somente para os índices em comum em ambos os arquivos. Como temos três datasets, usaremos o pipe para fazer a operação em um único fluxo de código.
dados_nelore <- inner_join(pn,p365, by = join_by(ID)) |>
inner_join(p550, by = join_by(ID))
dados_nelore |>
head()# A tibble: 6 × 4
ID PN P365 P550
<chr> <dbl> <dbl> <dbl>
1 RV143 36.0 211. 290.
2 YB821 31.1 195. 304.
3 QA008 32.3 187. 306.
4 WT997 28.4 192. 285.
5 IT185 25.1 202. 332.
6 PD544 37.5 190. 273.Parece confuso, mas é bem simples. Vamos por partes.
inner_join(pn,p365, by = join_by(ID)) : primeiro passamos os nossos conjuntos de dados como argumentos para a função inner_join, e com o argumento by fazemos a definição de qual variável será nossa chave de identificação ou índice, neste caso com by = join_by(ID) estamos informando que a coluna ID é a chave.
Ao fazermos isso já criamos um novo objeto que é a junção dos dados de PN e P365, e como mencionamos anteriormente, o |> lê sempre da esquerda para a direita, assim o nosso novo conjunto de dados do merge se torna a entrada da próxima função
inner_join(p550, by = join_by(ID)) : aqui o raciocínio é o mesmo, com a diferença de que como estamos no fluxo do pipe, o primeiro argumento é omitido pois o R já sabe que esse argumento vem do pipe anterior, e informamos então o conjunto de dados restante p550, a chave continua a mesma. E assim nosso merge está completo!
Existem outros tipos de joins no dplyr e você pode como sempre consultar a documentação para saber os detalhes e diferenças entre eles e quando utilizar cada tipo.
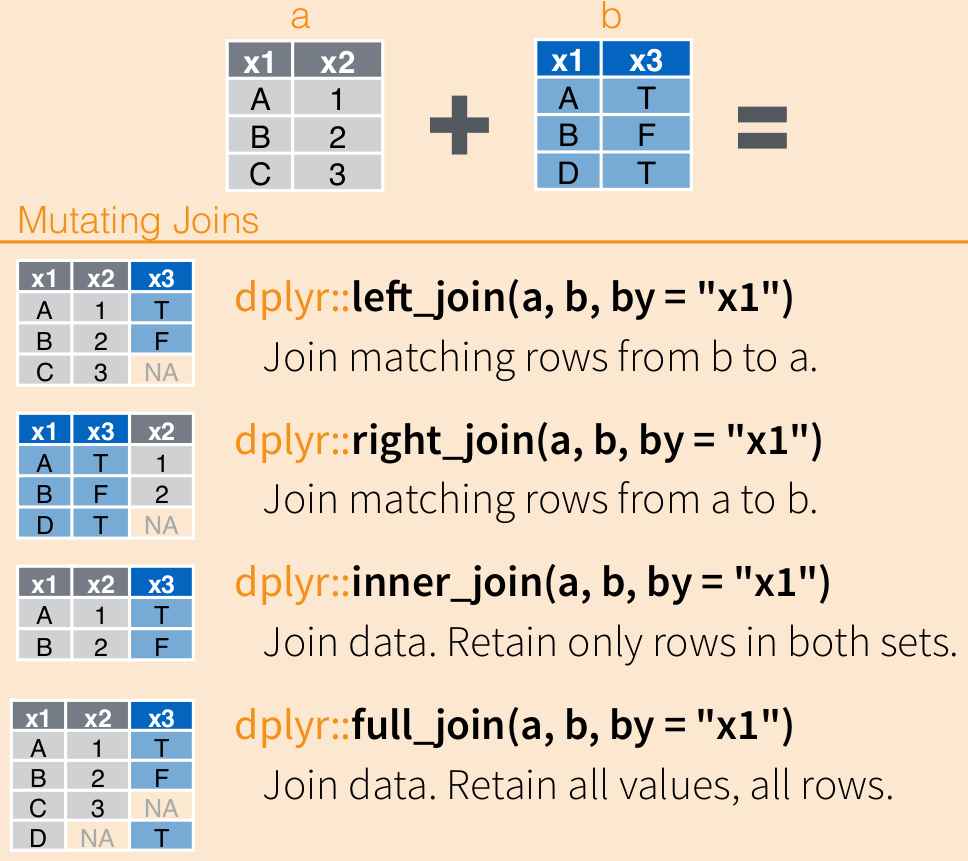
1.2 Modo Otimizado
No nosso exemplo temos somente três datasets que desejamos unir, mas existem situações aonde o número de datasets pode ser grande bem como o volume de dados contidos neles. Nessas situações podemos lançar mão de funções otimizadas para realizar tarefas repetitivas. A função map do pacote purrr é nossa amiga!
Vamos realizar a mesma operação do modo anterior mas de uma maneira mais eficiente tanto do ponto de vista de código quanto de gerenciamento de recursos do computador.
nelore <- list.files(path = "data",
pattern = "NEL.*.csv",
full.names = TRUE) |>
map(read_csv) |>
reduce(inner_join, by = join_by(ID)) |>
select(ID,PN,P365,P550)Rows: 50 Columns: 2
── Column specification ────────────────────────────────────────────────────────
Delimiter: ","
chr (1): ID
dbl (1): P365
ℹ Use `spec()` to retrieve the full column specification for this data.
ℹ Specify the column types or set `show_col_types = FALSE` to quiet this message.
Rows: 50 Columns: 2
── Column specification ────────────────────────────────────────────────────────
Delimiter: ","
chr (1): ID
dbl (1): P550
ℹ Use `spec()` to retrieve the full column specification for this data.
ℹ Specify the column types or set `show_col_types = FALSE` to quiet this message.
Rows: 50 Columns: 2
── Column specification ────────────────────────────────────────────────────────
Delimiter: ","
chr (1): ID
dbl (1): PN
ℹ Use `spec()` to retrieve the full column specification for this data.
ℹ Specify the column types or set `show_col_types = FALSE` to quiet this message.# A tibble: 6 × 4
ID PN P365 P550
<chr> <dbl> <dbl> <dbl>
1 RV143 36.0 211. 290.
2 YB821 31.1 195. 304.
3 QA008 32.3 187. 306.
4 WT997 28.4 192. 285.
5 IT185 25.1 202. 332.
6 PD544 37.5 190. 273.Vamos por parte novamente!
O primeiro passo foi gerar uma lista de arquivos a serem lidos automaticamente usando a função list.files. Com o argumento pattern = "NEL.*.csv" nós informamos que queremos listar somente os arquivos que comecem com NEL e sejam .csv. E por fim usamos full.names = TRUE para que ele nos retorne o caminho completo do arquivo incluindo o diretóriom p.e. data/NELPN.csv.
Com isso passamos o nosso vetor contendo os nomes dos arquivos para a função map. Esta função recebe uma lista/vetor e caminha pelos itens desta lista realizando a operação solicitada para cada item, no nosso caso irá executar a função read_csv para cada nome de arquivo informado no nosso vetor.
A função map retorna uma lista por padrão, no nosso caso uma lista de data.frame. Por isso ao final invocamos a função reduce para reduzir a nossa lista aplicando a função inner_join da mesma maneira que fizemos no modo anterior.
Neste exemplo pode parecer que o Modo 1 tem menos linhas de código e seja mais fácil, e de fato é. Entretanto, imagine se precisássemos ler 50 arquivos ao invés de três? Precisariamos criar 50 objetos e fazer o join individualmente destes, o que aumentaria substancialmente o número de linhas e também a quantidade de memória utilizada na operação.
2 Transformando Dados
Uma outra operação muito comum em edição de dados é transformar o formato do conjunto de dados entre formato longo (long) e formato largo (wide). Para demonstrar isso vamos recuperar nosso dataset de galinhas.
Attaching package: 'janitor'The following objects are masked from 'package:stats':
chisq.test, fisher.test# A tibble: 6 × 6
rep dieta_a dieta_b dieta_c dieta_d dieta_e
<dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 1 179 309 243 423 368
2 2 160 229 230 340 390
3 3 136 181 248 392 379
4 4 227 141 327 339 260
5 5 217 260 329 341 404
6 6 168 203 250 226 318Este dataset contem peso de galinhas submetidas a diferentes dietas. Como podemos observar ele se encontra no formato que chamamos de largo (wide), uma vez que cada dieta que seria o nosso tratamento está representada em uma coluna separada.
Na filosofia do tidyverse, para um dataset ser considerado tidy ou “arrumado”, cada célula deve ser uma observação completa. Para isso podemos transformar o nosso formato para formato longo (long).
gal_long <- galinhas |>
pivot_longer(dieta_a:dieta_e, names_to = "dieta", values_to = "peso") |>
arrange(dieta)
gal_long |>
head()# A tibble: 6 × 3
rep dieta peso
<dbl> <chr> <dbl>
1 1 dieta_a 179
2 2 dieta_a 160
3 3 dieta_a 136
4 4 dieta_a 227
5 5 dieta_a 217
6 6 dieta_a 168A função pivot_longer recebe um intervalo de colunas dieta_a:dieta_e no nosso caso, e transforma em uma nova variável que chamamos de dieta e os valores em uma outra chamada peso. Por fim usamos a função arrange para ordenar nossos dados pela coluna da dieta.
Também podemos fazer o caminho inverso agora.
# A tibble: 6 × 6
rep dieta_a dieta_b dieta_c dieta_d dieta_e
<dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 1 179 309 243 423 368
2 2 160 229 230 340 390
3 3 136 181 248 392 379
4 4 227 141 327 339 260
5 5 217 260 329 341 404
6 6 168 203 250 226 318Aqui a função pivot_wider recebe uma coluna com valores categóricos e cria colunas individuais para cada valor na variável, associando o valor de peso correspondente. Desta forma recuperamos o formato wide que existia nos dados originais.
